KT Aivle School 에이블스쿨 기자단] 8.28(월) ~ 9.3(일) 서울교육공모전 마무리 & 데이터분석 & 교육선발
이번 주의 스케줄
데이터 분석 수업 --> ADsP 내용과 겹치는 부분이 있어서 이해에 도움이 되었다
서울교육공모전 마무리
현재까지의 셀프 테스트 점수
교육 선발, 아쉽게 회식은 불참ㅠ
코딩 복습
이번주 후기 데이터 분석 수업으로만 이루어진 주였다. 핵심 개념 : 카이제곱검정, t검정, anova 분석, 피어슨 상관분석에서 p-value 해석! p-value 0.05 미만 일 때 채택하는 대립가설은 어떤 것인가
p-value 0.05가 기준선임은 확실히 기억해두면, 다른 부분은 헷갈릴 때 귀무가설이 무엇이지만 확인하면 손쉽게 문제를 해결할 수 있을 거라 생각한다
이번 주 수업 때마다 복습했던 CRISP-DM
드디어 수업과 병행하던 공모전을 마무리 했다!! 그동안 "수업 중간 쉬는 시간 & 점심 시간"에도 공모전 준비에 시간을 투자해서 정말 힘들었는데 이제 다시 쉬는 시간에는 쉴 수 있을 거 같다 휴..
첫 공모전 마무리!!!
현재까지의 셀프 테스트 점수 100점 흐름은 계속 유지하자!!
교육선발~~ AIVLE School에서 머신러닝 교육에 들어가기 미리 들어두면 좋을 거 같아 신청해둔 교육에 선발되었다
지금 코딩이 거의 노베이스 상태라 앞으로 참여할 수 있는 교육이 있다면 적극적으로 참여하려고 한
교육선발
이번주 프로그래머스 문제풀이 매주 일요일 내가 리더로 진행하는 스터디를 통해 꾸쭌히 프로그래머스 문제를 풀어가는 것이 큰 도움 이 된다고 느끼고 있다.
참여원이면 가끔 불참했을 수도 있는데 리더라 한 주도 빠지지 못하니 강제성 이 부여되어 더 열심히 할 수 있다고 느낀다.
돌아가며 자신들의 코드를 발표하는데, 다른 사람 코드를 보고 설명을 들으니 시야가 넓어지는 느낌도 받고 있다.
이번주 코딩 복습!!
컬럼 정보 데이터프레임.columns
컬럼 이름만 리스트에 담아 조회 list( 데이터프레임.columns )
데이터프레임 조건 조회(loc) 데이터프레임.loc [ (데이터프레임['컬럼명']==1) & (데이터프레임['컬럼명'] <=10) ]
데이터프레임 조건 조회 male_age = 데이터프레임.loc[ 데이터프레임['컬럼명'] == '원하는 데이터', '컬럼명']
결측치가 아닌 값 조회 데이터프레임.loc[ 데이터프레임['컬럼명'].notna() ]
특정 수치 사이값 조회 (ex) 10 ~ 20 사이 값 데이터프레임.loc[ 데이터프레임['컬럼명'].between(10, 20) ]
데이터프레임.loc[ (데이터프레임['컬럼명']>=10) & (데이터프레임['컬럼명'] <=20) ]
날짜 데이터 조회 데이터 프레임.loc['컬럼명'].isin( [ '날짜', '날짜' ] )
데이터프레임 값 변경 데이터프레임['컬럼명'] = 데이터프레임['컬럼명'].map({"원본 데이터" : "바꿀 데이터",
데이터 값 변경 pd.cut 데이터프레임['컬럼명'] = pd.cut(데이터프레임['컬럼명'], bins=[-np.inf, 30, 100, np.inf],
데이터 값 변경 np.where 데이터프레임['컬럼명'] = np.where(데이터프레임['컬럼명'] =='데이터값', 0, 1)
데이터프레임['컬럼명'] = 데이터프레임['컬럼명'].replace({'데이터값': 0, '데이터값 : 1})
데이터 합치기 Join, merge pd.merge( 데이터프레임, 데이터프레임, on ='컬럼명', how = 'left')
정렬 sort_values 데이터프레임.sort_values('컬럼명', ascending = False)
날짜 데이터
날짜 형식으로 변경 데이터프레임['컬럼명'] = pd.to_datetime(데이터프레임['컬럼명'])
연 데이터 추가 데이터프레임['Year'] = 데이터프레임['컬럼명'].dt.year
월 데이터 추가 데이터프레임['Month'] = 데이터프레임['컬럼명'].dt.month
단변량 분석_숫자형
평균 np.mean(데이터프레임['컬럼명'])
중앙값(중위수) 데이터프레임['컬럼명'].mean()
np.median(데이터프레임['컬럼명'])
최빈값 데이터프레임['컬럼명'].mode()
기초 통계량 전체 출력 데이터프레임.describe(include='all')
숫자형 시각화
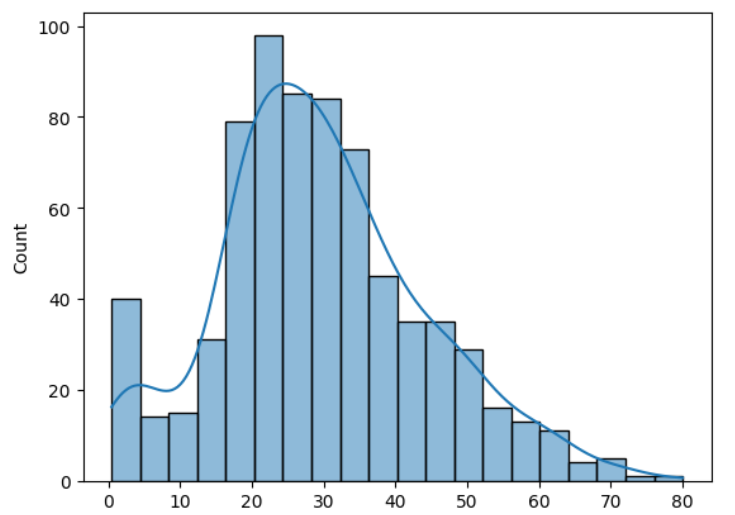
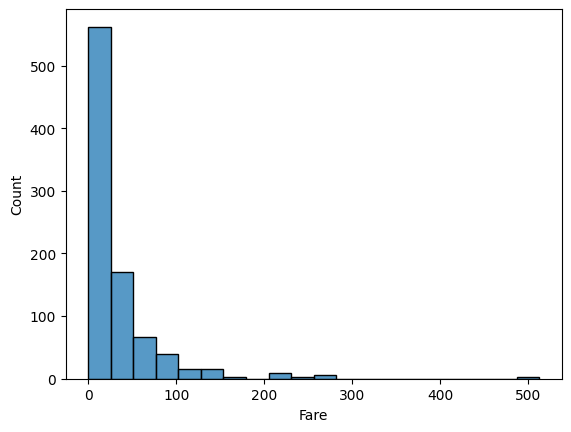
히스토그램
plt.hist(데이터프레임.컬럼명, bins= 갯수, edgecolor = '색상')
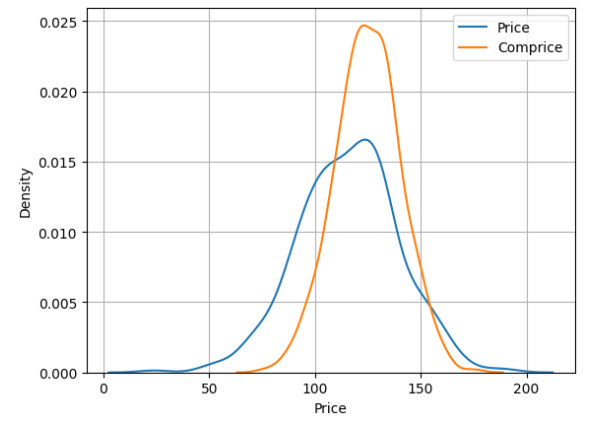
밀도함수
sns.kdeplot(데이터프레임['컬럼명'])
히스토그램 & 밀도함수 함께 표기
sns.histplot(데이터프레임['Age'], kde = True)
박스 플랏
plt.boxplot(데이터프레임['컬럼명'])
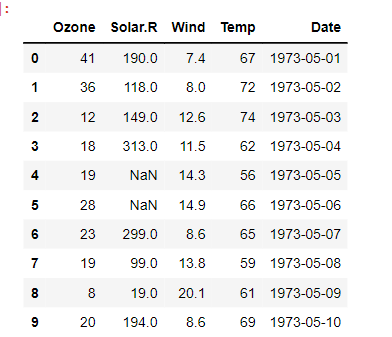
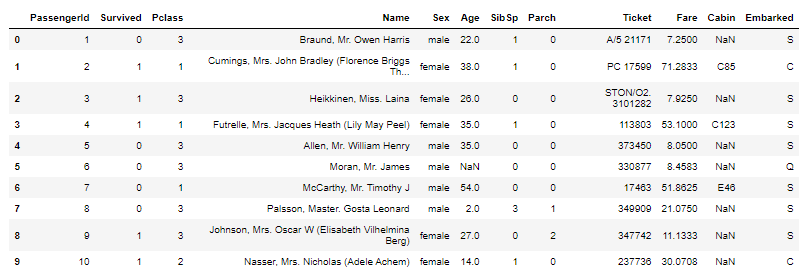
데이터 원본
단변량분석_범주형변수
범주별 빈도수 value_counts() : 범주의 개수와 상관 없이 범주별 개수를 계산
범주별 비율 계산(응용) 데이터프레임['컬럼명'].value_counts() / 데이터프레임.shape[0]
카운트 플랏 sns.countplot
sns.countplot( y='컬럼명', data= 데이터프레임)
sns.countplot( x='컬럼명', data= 데이터프레임)
기초 통계량 계산 0과 1 데이터 데이터프레임['컬럼명'].value_counts()
시각화 - 파이차트
plt.데이터프레임(컬럼명.values, labels = 컬럼명.index, autoptc='%.2%%',
원본 데이터
원본 데이터
이변량_숫자 vs 숫자
시각화 산점도
sns.scatterplot( x = '컬럼명', y = '컬럼명', data = 데이터프레임 )
pairplot으로 한 번에 시각화
sns.pairplot( 데이터프레임, kind = 'reg' )
jointplot
sns.pariplot( x='컬럼명', y= ' 컬럼명', data = 데이터프레임)
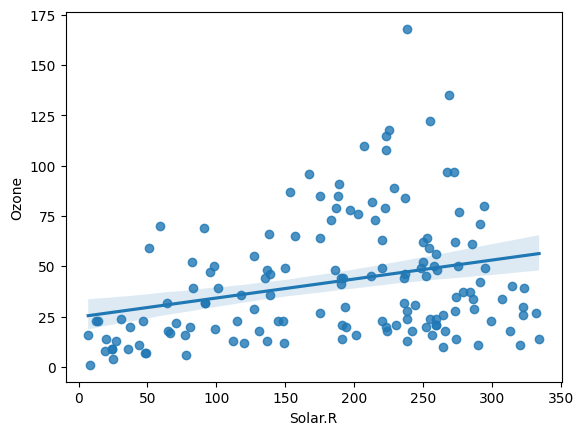
regplot
sns.regplot(x='컬럼명', y = '컬럼명' , data = 데이터프레임)
원본
이변량_숫자 vs 숫자 -> 상관분석
패키지 import scipy.stats as spst
상관계수와 p-value 계산 코
상관계수 구하기 데이터프레임.corr()
상관계수 히트맵 시각화
sns.heatplot( air.corr(),
평균 개념
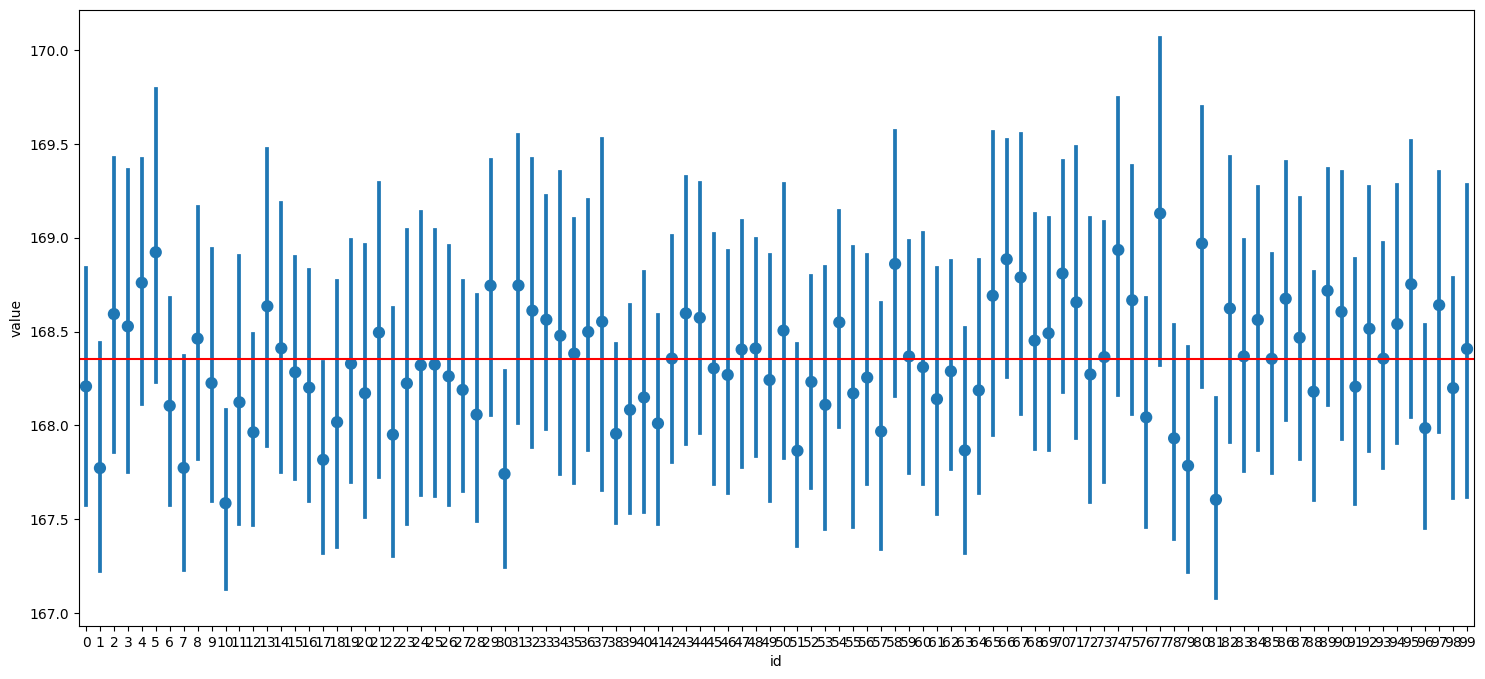
표준오차 SE Standard Error 표준오차는 표준편차와 다른 개념
95% 신뢰구간
sns.hisplot( 리스트, bins = 숫자)
errorbar
# 100번 샘플링
이변량_범주 vs 숫자
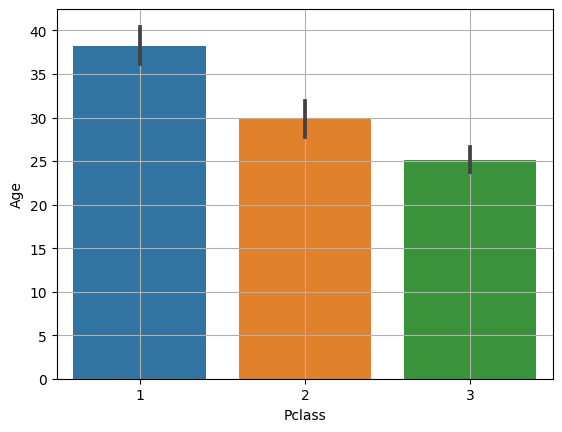
평균 비교 barplot
sns.barplot( x= ' 컬럼명', y = '컬럼명', data = 데이터프레임 )
boxplot sns.boxplot( x='컬럼명', y='컬럼명', data = 데이터프레임 )
NaN 결측치 제거 데이터프레임.loc [ 데이터프레임 ['컬럼명].notnull() ]
t-test 실제 코드
실제 코드 ( 성별에 대해 시행 )
t-test를 통해 얻은 p-value 값이 0.05 보다 크다면 두 집단 간의 평균에 큰 차이가 없다는 귀무가설을 채택한다.
anova Analysis Of VAriance
여러 집단 간에 차이 비교
원본 데이터
이변량 범주 vs 범주
교차표 pd.crosstab(행, 열) pd.crosstab(행, 열)
시각화 mosaic
mosaic( 데이터프레임, ['컬럼명', '컬럼명'] )
카이제곱 검정 범주형 변주들 사이에 어떤 관계가 있는지 수치화 카이제곱 검정 ttest
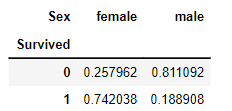
크로스탭 pd.crosstab(titanic['Survivde], titanic['Sex], normalize = 'columns')
이변량 숫자 vs 범주
숫자 --> 범주 시각화
sns.histplot( x = '숫자 컬럼', data = 데이터프레임, hue ='범주 컬럼' )
kdeplot 작성
sns.kdeplot( x='숫자 컬럼', data= 데이터프레임, hue = '범주 컬럼' )